


作者: 发布时间:2022-03-15 浏览次数:142
特邀:海南大学南海海洋资源利用国家重点实验室林桓课题组
林桓,海南大学南海海洋资源利用国家重点实验室副研究员,海南省领军人才,《Marine Resource and Ocean Science》编委,曾任岛津制作所全球应用技术开发中心副主任研究员。主要研究领域是基于高分辨以及定量质谱的核酸修饰生理学意义挖掘,在《Nature Communications》,Nucleic Acid Research等重要期刊发表过论文。
海南大学林桓副研究员团队以模式蓝藻(细长聚球藻 PCC 7942)为研究对象,与岛津广州分析中心展开深入合作,通过微流液相色谱仪(岛津Nexera Mikros)结合三重四级杆质谱仪(岛津LCMS-8050)建立了一套兼具灵敏度与特异性的修饰核苷鉴定方法,并对细长聚球藻PCC 7942 总tRNA组分中存在的修饰核苷进行了检测分析,为修饰核苷的鉴定提供了一种新思路。成果于2021年7月发表在《Journal of Separation Science》。

生物体中的RNA元件,包括核糖体RNA,信使RNA,转运RNA,剪接体RNA,miRNA等,均需要经历转录后修饰成为成熟的分子。这些修饰对实现RNA元件的功能至关重要,例如新冠病毒mRNA疫苗必须在RNA链上掺入修饰核苷才能在人体中稳定表达。RNA转录后修饰主要发生在核糖核苷(A/U/C/G)的碱基或核糖上,以共价结合的方式连接一个或多个化学基团。近年研究表明RNA修饰种类和修饰率的变化参与到RNA代谢及功能实现的全过程,具有重要生理学意义[1-5]。

分析方法灵敏度提高有三种途径:
目前质谱仪因其通用性高、选择性好和灵敏度高,是主流的检测技术,其中,灵敏度是评价质谱等级的重要指标之一。质谱仪的灵敏度受多方面影响,其中可以通过降低进入离子源的液流,有效提高离子化效率,从而较大提高质谱检测灵敏度。如目前比较常见的Nano液相、Micro液相和Semi-micro常规分析型液相,我们来做一下性能对比。
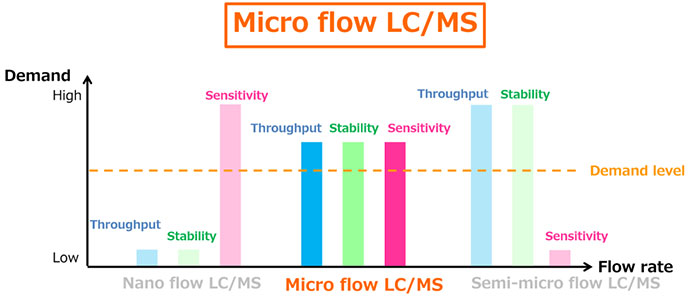
三种液相系统的性能对比
Nano液相的液量有利于LCMSMS离子源的去溶剂化,从而提高质谱响应。但是由于液流过低,其通量较小;另外,纳升级的流速对输液泵的精度和脉冲控制要求非常高,再加上其精密的部件对使用和维护都有很高的要求。故现有技术下Nano液相的通量和稳定性无法达到常规分析型液相的水平。而Semi-micro常规型液相已成为相对成熟、应用范围相对广的分析手段,这两项指标非常优异,但是,由于较大的液流,导致进入离子源,尤其是ESI离子源之后,其去溶剂化效率较差,灵敏度无法进一步提高。 Micro液相的色谱柱内径介于0.1~1.0 mm之间,流速一般介于1~100 μL之间,这一流速下的仪器稳定性、耐用性和维护性都比Nano液相有大幅提高,通量接近半微量分析型液相,而其去离子效率相比半微量分析型液相大大提高。因此Micro液相没有明显的短板,理论上是三种液相中相对理想的技术。

岛津微流量液相质谱联用系统 Mikros-LC+LCMS-8060
在本研究中研究人员报道了尿苷及其衍生物在低温及高有机溶剂的条件下容易析出造成信号变动,指出了利用HILIC等亲水原理的核苷酸LCMS分析中需要注意的问题。利用岛津微流量液相质谱联用系统测试了总tRNA组分中24个核苷标样,确定了各个标样所用的母离子和子离子,以及在HILIC色谱柱中的保留时间。通过多反应监测及保留时间,可以区分这些修饰核苷及其同分异构体。
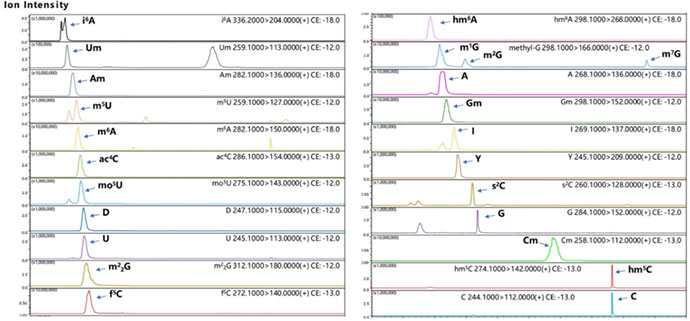
核苷标样微流量液质系统色谱图
测定了24个修饰核苷标样的最小检出量,运用微流液相色谱—三重四极杆质谱联用仪检出下限为0.1-1 fmol,而一般用于鉴定修饰核苷所用的半微流液相色谱—三重四极杆质谱联用仪的检出下限为1-10 fmol。
运用上述建立的方法,对细长聚球藻 PCC 7942中的修饰核苷进行定量检测,仅需含25 ng总tRNA组分的样品,即可得到清晰的质谱结果,而在传统的半微流液相色谱—三重四极杆质谱联用仪上得到相同结果,至少需要含100 ng的总tRNA组分的样品[6, 7]。
细长聚球藻样品图
海南大学林桓副研究员:修饰核苷中尿嘧啶核苷及其衍生物电离困难及各修饰核苷同分异构体之间不易区分的特点一直是质谱检测的难点。同时常规的半微流液相色谱—三重四极杆质谱联用仪检出限较高,需要的样品量较大,这也是不易提取的样品检测的制约条件之一。该研究依托岛津公司强大的质谱分析平台,首次使用微流液相色谱—三重四极杆质谱联用仪分离鉴定总tRNA组分中的修饰核苷,灵敏度较半微流液相色谱——三重四极杆质谱联用仪提高了10倍以上。该方法可支持表观转录组学的发展。
【参考文献】
[1] Lin H, Miyauchi K, Harada T, Okita R, Takeshita E, Komaki H, Fujioka K, Yagasaki H, Goto Y-i, Yanaka K, Nakagawa S, Sakaguchi Y, Suzuki T. CO2-sensitive tRNA modification associated with human mitochondrial disease. Nature Communications. 2018; 9: 1875.
[2] Lian H, Wang Q H, Zhu C B, Ma J, Jin W L. Deciphering the Epitranscriptome in Cancer. Trends in Cancer. 2018: S2405803318300219.
[3] Schimmel, Paul. The emerging complexity of the tRNA world: mammalian tRNAs beyond protein synthesis. Nature Reviews Molecular Cell Biology. 2017.
[4] Thomas J M, Batista P J, Meier J L. metabolic Regulation of the Epitranscriptome. Acs Chemical Biology. 2019.
[5] Chionh Y H, Mcbee M, Babu I R, Hia F, Lin W, Zhao W, Cao J, Dziergowska A, Malkiewicz A, Begley T J. tRNA-mediated codon-biased translation in mycobacterial hypoxic persistence. Nature Communications. 2016; 7: 13302.
[6] Kimura S, Dedon P C, Waldor M K. Comparative tRNA sequencing and RNA mass spectrometry for surveying tRNA modifications. Nature Chemical Biology. 2020.
[7] Su D, Chan C T Y, Gu C, Lim K S, Chionh Y H, Mcbee M E, Russell B S, Babu I R, Begley T J, Dedon P C. Quantitative analysis of ribonucleoside modifications in tRNA by HPLC-coupled mass spectrometry. Nature Protocols. 2014; 9: 828-841.
本文内容非商业广告,仅供专业人士参考。